K-分割交差検証
前回記事で扱ったホールドアウト検証は、入力データと目標データを一つの方法で分割するので、結果に偏りが生じる可能性があります。そこで今回は下図のようにデータを 4 等分して、テストデータと訓練データの役割を変えながら、近似曲線とテストデータの誤差を計算し、最後に 4 個の標準偏差の平均を求めて、これをモデルの評価基準とします。
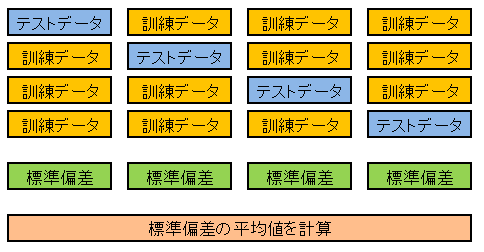
標準偏差の平均が最小となる基底関数の組合わせが決定したら、すべての入力データ x と目標データ y を使ってパラメータベクトルを計算し直して、これを最終的なモデルとして採用します。
一般に、データを K 分割して誤差を平均して、ばらつきの影響を軽減する手法を K-分割交差検証 (K-fold cross validation) といいます。
上図のようにデータを分割する準備として、x のインデックス配列を作成しておきます。
# In[13] # xのインデックス配列 idx = np.arange(0, x.shape[0]) print(idx) # [0 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19]
この配列を 4 で割ったときに、割り切れない要素と割り切れる (余りが 0 となる) 要素に分けると、配列を大きさ 3 : 1 の比で分割できます。
# In[14]
# 4で割って余りが0以外の要素
idx_train = idx[np.fmod(idx, 4) != 0]
# 4で割って余りが0になる要素
idx_test = idx[np.fmod(idx, 4) == 0]
print("idx_train:\n{}".format(idx_train))
print("idx_test:\n{}".format(idx_test))
# idx_train:
# [ 1 2 3 5 6 7 9 10 11 13 14 15 17 18 19]
# idx_test:
# [ 0 4 8 12 16]
マスク操作によって、入力データ x を分割できます。
# In[15]
# 入力データxを分割
x_train = x[idx_train]
x_test = x[idx_test]
print("x_train:\n{}".format(x_train))
print("x_test:\n{}".format(x_test))
# x_train:
# [16 22 43 66 20 13 1 39 62 33 8 28 24 18 3]
# x_test:
# [35 5 52 45 71]
より一般的な分割に対応できるように、train_test() 関数を定義しておきます。
# In[16]
# 学習用データとテストデータに分ける関数
def train_test(x, y, k, r):
idx = np.arange(0, x.shape[0])
idx_test = idx[np.fmod(idx, k) == r]
idx_train = idx[np.fmod(idx, k) != r]
x_test = x[idx_test]
x_train = x[idx_train]
y_test = y[idx_test]
y_train = y[idx_train]
return x_train, x_test, y_train, y_test
train_test() の x, y にはそれぞれ入力データと目標データを渡します。k には分割数、r には剰余 (remainder) を渡します。戻り値は x, y それぞれの学習用データとテストデータです。たとえば、データを 4 で割ったとき、剰余 1 で分類するには次のように記述します。
# In[17]
# データを4で割ったときの剰余1で分割
x_train, x_test, y_train, y_test = train_test(x, y, 4, 1)
print("x_train:\n{}".format(x_train))
print("x_test:\n{}".format(x_test))
print("y_train:\n{}".format(y_train))
print("y_test:\n{}".format(y_test))
# x_train:
# [35 22 43 5 20 13 52 39 62 45 8 28 71 18 3]
# x_test:
# [16 66 1 33 24]
# y_train:
# [85.19 64.27 68.91 21.27 60.07 55.18 88.08 82.31
# 81.18 80.76 37.55 75.9 72.39 62.04 12.47]
# y_test:
# [58.93 68.88 8.89 78.98 69.51]
インデックス配列を 4 で割ったときに
・余りが 0 にならない要素と、0 となる要素
・余りが 1 にならない要素と、1 となる要素
・余りが 2 にならない要素と、2 となる要素
・余りが 3 にならない要素と、3 となる要素
のように分けると、データを 4 通りの方法で分割できます。
# In[18]
# データを剰余0,1,2,3で分割
for r in range(4):
x_train, x_test, y_train, y_test = train_test(x, y, 4, r)
print("r = {}".format(r))
print("x_train:\n{}".format(x_train))
print("x_test:\n{}".format(x_test))
print("y_train:\n{}".format(y_train))
print("y_test:\n{}\n".format(y_test))
'''
r = 0
x_train:
[16 22 43 66 20 13 1 39 62 33 8 28 24 18 3]
x_test:
[35 5 52 45 71]
y_train:
[58.93 64.27 68.91 68.88 60.07 55.18 8.89 82.31
81.18 78.98 37.55 75.9 69.51 62.04 12.47]
y_test:
[85.19 21.27 88.08 80.76 72.39]
r = 1
x_train:
[35 22 43 5 20 13 52 39 62 45 8 28 71 18 3]
x_test:
[16 66 1 33 24]
y_train:
[85.19 64.27 68.91 21.27 60.07 55.18 88.08 82.31
81.18 80.76 37.55 75.9 72.39 62.04 12.47]
y_test:
[58.93 68.88 8.89 78.98 69.51]
r = 2
x_train:
[35 16 43 5 66 13 52 1 62 45 33 28 71 24 3]
x_test:
[22 20 39 8 18]
y_train:
[85.19 58.93 68.91 21.27 68.88 55.18 88.08 8.89
81.18 80.76 78.98 75.9 72.39 69.51 12.47]
y_test:
[64.27 60.07 82.31 37.55 62.04]
r = 3
x_train:
[35 16 22 5 66 20 52 1 39 45 33 8 71 24 18]
x_test:
[43 13 62 28 3]
y_train:
[85.19 58.93 64.27 21.27 68.88 60.07 88.08 8.89
82.31 80.76 78.98 37.55 72.39 69.51 62.04]
y_test:
[68.91 55.18 81.18 75.9 12.47]
'''
次に、学習データとテストデータを与えて、テストデータに対する標準偏差を計算する関数を定義します。
# In[19]
# テストデータの標準偏差を計算する関数
def std_test(x_train, x_test, y_train, y_test, func):
z = Fit_func(x_train, y_train, func)
mse = np.mean((z.line(x_test)-y_test)**2)
return np.sqrt(mse)
適当な基底関数をセットして、テストデータの標準偏差を計算してみます。
# In[20]
# 基底関数をセット
basis = gauss_basis(5, 10)
basis.append(lambda x:1)
basis.append(lambda x: np.log(1+x))
# 学習用データでフィッティング
z = Fit_func(x_train, y_train, basis)
# 回帰曲線とテストデータの標準偏差
sd_test = std_test(x_train, x_test, y_train, y_test, basis)
print("{:.3f}".format(sd_test))
# 7.928
分割の仕方を変えながらテストデータの標準偏差を計算し、平均値を返す k_fold() 関数を定義します。
# In[21]
# K分割交差検証関数
def k_fold(x, y, k, func):
sd = 0
for r in range(k):
x_train, x_test, y_train, y_test = train_test(x, y, k, r)
sd += std_test(x_train, x_test, y_train, y_test, func)
return sd / k
k_fold() を使って、標準偏差の平均値が最小となるようなガウス基底の数を探します。
# In[22]
# ガウス基底の数を格納するリスト
n = []
# 標準偏差の平均値を格納するリスト
sd_list = []
# ガウス基底の数ごとに標準偏差の平均値を計算
for i in range(7):
basis = gauss_basis(i, 10)
basis.append(lambda x:1)
basis.append(lambda x: np.log(1+x))
sd = k_fold(x, y, 4, basis)
n.append(i)
sd_list.append(sd)
# ガウス基底の数と標準偏差の平均値をプロット
fig = plt.figure(figsize=(6, 4))
ax = fig.add_subplot(111)
ax.grid()
ax.set_xlabel("Number of Gaussian basis", fontsize=14)
ax.set_ylabel("SD_test_avg", fontsize=14)
ax.plot(n, sd_list, color="green")
plt.show()
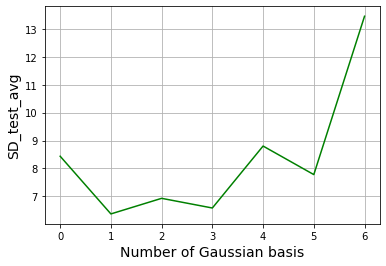
ガウス基底の数が 1 のときに標準偏差の平均値は最小となります。前回のホールドアウト検証で得た結果と同じく、ガウス関数、対数関数、定数関数の 3 個の基底で表される比較的単純なモデルが最良だということです。最後に全てのデータを使ってパラメータを最適化します。
# In[23]
# 基底をセット
basis = gauss_basis(1, 10)
basis.append(lambda x:1)
basis.append(lambda x: np.log(1+x))
# フィッティング
z = Fit_func(x, y, basis)
# 回帰曲線データ
x2 = np.linspace(0, 70, 100)
y2 = z.line(x2)
# データのプロット
fig = plt.figure(figsize=(6, 4))
ax = fig.add_subplot(111)
ax.grid()
ax.set_title("Regression curve", fontsize=16)
ax.set_xlabel("Age", fontsize=14)
ax.set_ylabel("Weight [kg]", fontsize=14)
ax.scatter(x, y, color="blue")
ax.plot(x2, y2, color="red")
plt.show()

年齢と体重の関係が湾曲部のない曲線となることは直感的にも納得できます。この種の曲線を表現するときに、対数関数が大きな役割を担います。ガウス関数だけの基底だと、もっと多くの関数が必要となります。
コメント
下記は誤植と思われますので、ご確認ください。
In[17] プログラムのコメントで、5 で割った → 4 で割った
In[21] プログラムで、
z = Fit_func(x_train, y_train, func)
は、std_test( ) 関数内でも定義されているので不要だと思います。
修正しました。
z=Fit_func(x_train,y_train,func)
はまったく不要でした。申し訳ないです。